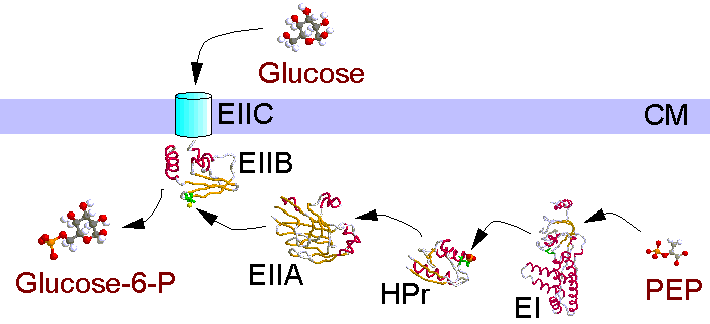
Das bakterielle Phosphotransferase-System ist für die spezifische Zuckeraufnahme in die Zellen zuständig, wobei die Zucker gegen einen Konzentrationsgradienten bei gleichzeitiger Phosphorylierung transportiert werden. Phosphatdonor ist das "energiereiche" Phosphoenolpyruvat (PEP). Über die löslichen Enzyme EI und HPr, die nicht zuckerspezifisch sind, wird das Phosphat auf einen Enzymkomplex EII übertragen. Dieser besteht aus den Komponenten A, B und C, wobei je nach Spezifität und Bakterium die Komponenten auch Domänen eines Proteins sein können. Komponente/Domäne C ist das eigentliche Transportprotein und in der Membran verankert. Im Glucose-PTS von E. coli ist EIIA ein lösliches Protein, EIIB/C ist membranständig.
Die vom PEP abgespaltene Phosphatgruppe wird in den Proteinen kovalent an Histidin oder Cystein gebunden. Der Phosphorylierungsgrad der Enzyme dirigiert auch Regelmechanismen in der Zelle (Katabolitrepression, Chemotaxis).
Enzym I wird durch PEP autokatalytisch phosphoryliert. Das 64 kD große Protein ließ sich bisher nicht kristallisieren, aber der aminoterminale Teil (inclusive des aktiven His) ist für eine Strukturanalyse zugänglich. In dem hier gezeigten Modell sind die Aminosäuren 2 - 249 sichtbar.