Botanik online 1996-2004. Die Seiten werden nicht mehr bearbeitet, sie bleiben als historisches Dokument der botanischen Wissenschaft online erhalten!
Chloroplasten sind die typischen Organellen grüner Pflanzen. In ihnen läuft die Photosynthese ab, doch findet man daneben eine Reihe weiterer synthetischer Leistungen.
Chloroplasten (und andere Plastiden, z.B Leukoplasten, Amyloplasten, Chromoplasten) entstehen durch Teilung auseinander, oder aus Proplastiden, einem Differenzierungsstadium, das bei der Geschlechtszellbildung entsteht, und aus denen sie unter Lichteinwirkung gebildet werden können.
In unbelichteten Plastiden formen die inneren Membranen einen gitterförmig strukturierten Prolamellarkörper aus, der bei Belichtung in das Thylakoidsystem übergeht. Bei einzelligen Algen (Euglena, Chlamydomonas) sowie bei höheren Pflanzen, z.B. der Gerste (Hordeum vulgare), sind eine Vielzahl von Mutanten beschrieben worden, bei denen die Synthese der Chloroplasten an verschiedenen Stellen blockiert ist. Durch das Studium solcher Mutanten gelang es, die einzelnen Schritte zu verfolgen, und die Teilabschnitte der Synthese in die richtige Reihenfolge zu bringen. In grünen Geweben stehen die Teilungsrate der Chloroplasten und die Mitoserate in enger Beziehung zueinander.
Im Gegensatz zu den bisher besprochenen Membranen und Kompartimenten sind Chloroplasten aus vielen Pflanzenarten leicht zu isolieren; in der Regel durch Dichtegradientenzentrifugation oder - wie früher - differentielle Zentrifugation. Daher wissen wir auch über deren Membranen und die Reaktionen im Kompartiment gut Bescheid.
Es gibt drei grundsätzlich voneinander verschiedene Membrantypen:
![]() Die äußere Membran. Sie ähnelt in ihrer Zusammensetzung
anderen cytoplasmatischen Membranen.
Die äußere Membran. Sie ähnelt in ihrer Zusammensetzung
anderen cytoplasmatischen Membranen.
![]() Die innere Membran, und schließlich
Die innere Membran, und schließlich
![]() die Thylakoidmembranen (Photosynthesemembranen).
die Thylakoidmembranen (Photosynthesemembranen).
Diagramm zur Charakterisierung der Membranoberflächen und -seiten nach Gefrierätzbehandlung. Die Bezeichnungen beruhen auf einer von BRANTON et al. vorgeschlagenen Nomenklatur. P: dem Protoplasma (Cytosol, Stroma) zugekehrte Membranoberfläche; E: dem exoplasmatischen Lumen zugekehrte Membranflächen; F: membraninterne Fraktur; S: Oberfläche (surface) der Membran; s: stacked, Membrananteile in gestapelten Membranen; u: unstacked, Membrananteile in nicht-gestapelten Membranen. (Nach L. A. STAEHELIN, 1976)
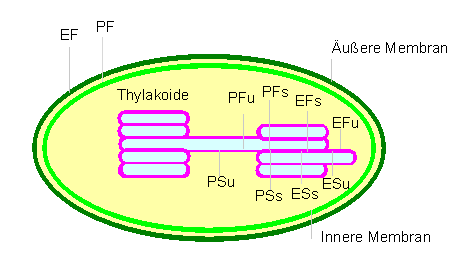
Die äußere und die innere Membran faßt man im allgemeinen
als Chloroplastenhülle zusammen. Thylakoidmembranen
entstehen durch Einstülpungen und anschließende Abschnürungen
der inneren Membran. Die Thylakoidmembranen wiederum trennt man in die
Grana- und die Stromalamellen (gestapelte und nichtgestapelte Bereiche).
Mehr über die Beteiligung der einzelnen Membranabschnitte an den Reaktionen
der Photosynthese:
In Zellen der Gefäßbündelscheide der sogenannten C4-Pflanzen (Zuckerrohr, Mais u.a) findet man unvollständige Chloroplasten mit nur Teilaktivitäten des Photosyntheseprozesses (PS I). Stroma- und Granabereiche können reversibel ineinander überführt werden. Das Fehlen einer Aktivität in Stromathylakoiden bedeutet jedoch nicht, daß die benötigten Enzyme fehlen. Es konnte nämlich gezeigt werden, daß sie auch dort vorhanden, doch nicht zu funktionellen Einheiten (oligomeren Komplexen) vereint sind. Die Aktivitäten werden also durch das Aggregationsverhalten der Untereinheiten und kooperative Wechselwirkung zwischen übereinander liegenden Membranen gesteuert.
Die chemische Zusammensetzung und die Eigenschaften der Chloroplastenmembranen wurden seit 1973 vorwiegend im Laboratorium von R. DOUCE analysiert. Sie enthalten ein Enzymsystem, das Galactosyleinheiten von Uridindiphosphatgalactose auf Diglyceride übertragen kann. Die dabei entstehenden Monogalactosyldiglyceride und Digalactosyldiglyceride werden in Chloroplastenmembranen eingebaut, sind deren typisches Kennzeichen. Die beiden äußeren Membranen enthalten darüber hinaus das Xanthophyll Violaxanthin, das ihnen eine schwach gelbliche Färbung verleiht. Stark belichtete Chloroplastenhüllen erscheinen mehr orange, was auf eine Umwandlung von Violaxanthin in Zeaxanthin zurückzuführen ist.
Chloroplasten enthalten genetische Information. Ihre Bedeutung als Erbträger geht auf frühe Arbeiten von E. BAUR (1909) und C. CORRENS (1909) zurück . Die molekulargenetische Phase der Forschung setzte 1962 mit dem definitiven Nachweis von DNS in Chloroplasten ein (H. RIS und W. PLAUT, University of Wisconsin, Madison). Das DNS-Molekül (ctDNS) ist zirkulär. Obwohl es die Information zur Bildung einer Reihe von Chloroplastenproteinen trägt, reicht die Information bei weitem nicht aus, um alle in Chloroplasten nachgewiesenen Proteine codieren zu können:
Zur Instruktion der DNS- und Proteinbiosynthese in Chloroplasten allein würde man 100 Gene benötigen (DNS-Polymerasen, RNS-Polymerasen, rRNS, ribosomale Proteine, tRNS, Aminoacyl-tRNS-Synthetasen, lösliche Faktoren). Hinzu kämen etwa 40 Enzyme, die für die Chlorophyll- und Carotinoidsynthese gebraucht werden sowie weitere 40 für die übrigen Aktivitäten der Photosynthese. Für diese Gene würde die Chloroplasten-DNS rein rechnerisch vielleicht gerade ausreichen. Bei nahezu allen höheren Pflanzen (soweit untersucht) enthält sie rund 150 000 Basenpaare. Doch ist die Liste der in Chloroplasten nachgewiesenen Aktivitäten damit immer noch nicht erschöpft. Es werden weiterhin benötigt:
![]() Enzyme für Lipidsynthesen und Aminosäuresynthesen,
Enzyme für Lipidsynthesen und Aminosäuresynthesen,
![]() Enzyme für den Kohlenhydratstoffwechsel, und schließlich
Enzyme für den Kohlenhydratstoffwechsel, und schließlich
![]() Enzyme für die Synthese einer Reihe sekundärer Pflanzenstoffe.
Enzyme für die Synthese einer Reihe sekundärer Pflanzenstoffe.
| C N O Cl |
Das allein macht deutlich, daß eine Anzahl von Enzymen aus dem Cytosol in die Chloroplasten importiert werden müssen, und das sind gar nicht so wenige. Es besteht ein diffiziles Gleichgewicht zwischen chloroplastencodierten und kerncodierten Funktionen. Die Chloroplasten-DNS wird repliziert, die dafür benötigten Enzyme sind kerncodiert, sie werden im Cytosol gebildet und anschließend in die Chloroplasten überführt. Die Chloroplastenribosomen enthalten rRNS, die chloroplastencodiert ist und Proteine, von denen einige chloroplastencodiert, andere wiederum kerncodiert sind. Die Entscheidung darüber, ob ein Protein im Cytosol oder in den Chloroplasten gebildet wird, läßt sich durch Einsatz spezifischer Inhibitoren (Antibiotika) fällen. Streptomycin und Chloramphenicol (Bild links) hemmen die Proteinbiosyntese an Chloroplastenribosomen (und Ribosomen der Prokaryonten). Cyoloheximid hat auf sie keinen Einfluß, hemmt dafür aber die Proteinbiosynthese an Ribosomen des Cytosols. Unter Einsatz dieser Hemmstoffe konnte die Herkunft und der Syntheseort einer Reihe von Proteinen ermittelt werden.
Ein weiterer wichtiger Zugang zur Klärung dieser Frage erfolgte über den Einsatz von Mutanten (z.B. von Euglena gracilis und Chlamydomonas reinhardii). Durch genetische Analyse kann entschieden werden, ob ein Defekt dem Chloroplasten- oder dem Kerngenom zuzuschreiben ist, und durch eine biochemische Analyse (Auftrennung der Proteine durch Gelelektrophorese und/oder isoelektrische Fokussierung) kann er auf Veränderung eines bestimmten Proteins zurückgeführt werden.
Ein dritter Zugang wäre schließlich die Analyse der Proteinbiosynthese in isolierten Chloroplasten und in in vitro - Proteinbiosynthesesystemen, die aus Chloroplastenbestandteilen aufgebaut werden können. Ein wichtiger Aspekt ist der Befund, daß die oligomeren Proteine der Photosynthesemembran aus Untereinheiten bestehen, die zum Teil chloroplastencodiert, zum Teil kerncodiert sind. Es sind also gute Beispiele zur Untersuchung der Wechselwirkung zwischen den beiden Genomen.
Ungeklärt bleibt dabei die Frage, wie sich eine solche Kooperation im Verlauf der Evolution herausgebildet hat. Gehen wir von der Endosymbiontenhypothese und einigen vergleichbaren Befunden der Molekularbiologie ("wandernde", "springende" Gene) aus, ist es durchaus plausibel anzunehmen, daß ursprünglich die gesamte benötigte genetische Information in der Chloroplasten-DNS lokalisiert war, daß dann aber Teile aus den Chloroplasten in den Kern einwanderten und dort seither als Kerngene repliziert und exprimiert werden. Zur Illustration der folgende Befund: Bei der Erbse (Pisum sativum) werden die gamma- und die delta- Untereinheit der ATPase im Cytosol synthetisiert, während die alpha-, beta- und epsilon-Untereinheiten in Chloroplasten gebildet werden (P.-Y. BOTHYETTE und A. T. JAGENDORF, 1978). In Spinatchloroplasten hingegen wird neben den drei letztgenannten Untereinheiten auch die gamma- Untereinheit in den Chloroplasten gebildet (N. NELSON, I. H. NELSON und G. G. SCHATZ, 1980).
Wie sehen die Transportmechanismen durch die Chloroplastenhülle aus? Ein im Cytosol gebildetes Protein muß mindestens zwei Membranen (äußere und innere Plastidenmembran) überwinden. Einige wenige, kerncodierte, dann aber an der Thylakoidinnenseite aktive Proteine (z.B. das Plastocyanin), müssen sogar drei Membranen passieren (S. SMEEKENS et al., 1985). An der äußeren Chloroplastenmembran findet man, zumindest bei höheren Pflanzen, keine Ribosomen. Der Mechanismus der Proteinsynthese durch die Membran hindurch kann also nicht zutreffen. Es muß folglich einen Transport bereits fertiger Proteine geben. B. DOBBERSTEIN, G. BLOBEL und N.-H. CHUA (Rockefeller University, 1977) fanden, daß die im Cytosol gefertigten, für den Transport in die Chloroplasten bestimmten Proteine um etliches größer als die späteren funktionellen Formen in Chloroplasten sind.
Den zusätzlichen Abschnitt belegte man mit dem Begriff Transitsequenz (= Transfersequenz). Sie muß offensichtlich Rezeptoren auf der Außenseite der Chloroplasten erkennen, und es muß spezifische Carrier geben, die die Proteine binden und durch die Membranen translocieren. Ferner wird eine Endoprotease benötigt, die die Proteinvorstufen (die Transportform) in die aktive Form bringt. Ein solches Enzym konnte aus Chloroplasten von Chlamydomonas reinhardii isoliert werden.
Eine Vorbehandlung von Chloroplasten mit einer Protease unterbindet jegliche Proteinaufnahme, d.h., die benötigten Rezeptoren für die Transitsequenzen und die Carrier wurden außer Kraft gesetzt, und ohne sie kann kein Protein eingeschleust werden.
Der beschriebene Mechanismus erklärt aber noch nicht, wie die innere Chloroplastenmembran überwunden wird. Doch das erwies sich als einfacher, als man sich ursprünglich vorgestellt hatte. R. DOUCE und Mitarbeiter fanden (1973), daß die Abstände der beiden Membranen der Chloroplastenhülle nicht konstant seinen, daß beide Membranen schwingen, daß eine oszillierende Bewegung entsteht, so daß sie sich in regelmäßigen Zeitabständen punktuell berühren und kurzfristig und reversibel fusionieren. An solchen Stellen hätte ein Protein nur eine Membran zu durchdringen.
Viele der anstehenden Probleme sind in den letzten Jahren mit Hilfe der Gentechnik angegangen worden. Dabei wurde u.a. auch gefunden, daß es eine sogenannte promiscuous DNA gibt. Man versteht darunter DNS-Abschnitte, die sowohl in Chloroplasten, Mitochondrien als auch im Kern vorkommen. Ferner wurde gezeigt, daß die DNS aus Mais-Mitochondrien das Gen der Ribulose-1,5-Bisphosphat-Carboxylase enthält (D. M. LONSDALE et al., 1983) und daß es Homologien zwischen ribosomaler und messenger-RNS aus Chloroplasten, Mitochondrien (und Escherichia coli) gibt (H. J. BOHNERT et al., 1980). Solche Befunde weisen auf einen Austausch genetischer Information zwischen den einzelnen Kompartimenten der Zelle hin.
Modell der Wechselwirkungen zwischen plastiden- und kerncodierten Transkriptions- und Translationsprodukten. TS: Transitsequenz, ein N-terminaler Abschnitt der Polypeptidkette, der für das Durchschleusen des Proteinmoleküls durch die Membran benötigt wird und der anschließend proteolytisch abgetrennt wird. (Nach W. BOTTOMLEY und H. J. BOHNERT, 1982).

Das bisher Vorgetragene sind Ergebnisse, die bis zum Zeitpunkt der vollständigen Sequenzierung der Chloroplasten-DNS zweier Pflanzenarten bekannt waren. Im Sommer 1986 wurden die Nukleotidsequenzen der Chloroplasten-DNS von Marchantia polymorpha (einem Lebermoos) und Nicotiana tabacum veröffentlicht. Zwei japanische Arbeitsgruppen (K. OHYAMA und 12 Mitarbeiter, Kyoto University, und K. SHINOZAKI und 22 Mitarbeiter, Nagoya University) vollbrachten das Werk. Die Marchantia-ctDNS enthält 121 024 Basenpaare, die Tabak-ctDNS 155 844. Trotz des nicht zu übersehenden Größenunterschiedes enthalten die Moleküle nahezu den gleichen Satz an Genen - in nahezu unveränderter Anordnung. Allein dieser Befund weist zwingend darauf hin, daß die beiden Chloroplastengenome auf einen gemeinsamen Vorfahren zurückzuführen sind. Das Marchantia-Chloroplastengenom trägt vermutlich 128 Gene, davon 4 rRNS-Gene, 32 tRNS-Gene und 55 sogenannte offene Leseraster (open reading frames, ORF). Offene Leseraster sind Nukleotidsequenzen, die mit einem Startcodon beginnen, einem Stoppcodon enden und von keinerlei derartigen Signalen unterbrochen sind. Vieles spricht dafür, daß solche Abschnitte Strukturgene für Proteine sind. Da man bei einer gegebenen Nukleotidsequenz auch die zugehörige Aminosäuresequenz aufschreiben kann, ließen sich etliche Proteingene identifizieren. So wurde z.B. ein Nukleotidabschnitt gefunden, der die Information für eine NADH-Dehydrogenase-Untereinheit enthält, die man bislang nur in menschlichen Mitochondrien nachgewiesen hatte; auch das ein Hinweis auf promiscuous DNA.
Die Chloroplasten-DNS ähnelt in ihrer Struktur einer Prokaryoten-DNS. So werden u.a. hintereinanderliegende Gene en bloc transkribiert (Konzept eines Operons). Ferner gibt es in einigen Fällen überlappende Gene, d.h., Nukleotidsequenzen, die zugleich Bestandteil zweier Gene sind. In einfacher Darstellung: Die Sequenz für Gen1 sei
A-B-C-D-E-F und für Gen 2 E-F-G-H-I...
Etwa 20 Gene enthalten Introns unterschiedlicher Länge; das wäre eine Eigenschaft der Eukaryoten-DNS.
Die Chloroplasten-DNS des Tabaks enthält das Gen für ein bestimmtes ribosomales Protein (S 16, d.h., Protein Nr. 16 der kleinen (small) ribosomalen Untereinheit); dieses Gen fehlt der Lebermoos-ct-DNS. Dafür enthält diese das Gen für ein anderes ribosomales Protein [L 21; also Protein Nr. 21 der großen (large) Untereinheit], welches wiederum der ctDNS aus Lebermoos fehlt.
Die Chloroplasten sind der Ort der Photosynthese und daher ist es auch wichtig, zu erkennen, welche Metaboliten von den Chloroplasten aufgenommen werden und welche an das umgebende Cytosol wieder abgegeben werden. Aufgenommen werden vornehmlich Wasser, Kohlendioxyd und Phosphat. Im Stroma der Chloroplasten laufen die Reaktionen des CALVIN-Zyklus ab. Das Endprodukt Glucose wird ins Cytosol transportiert, dort dient es u.a. der Synthese von Saccharose, die entweder in der Vakuole gespeichert wird oder aus der Zelle exportiert wird. Neben der Saccharose werden vorwiegend die Triosephosphate Dihydroxyacetonphosphat und Glycerinaldehydphosphat der Pflanzenzelle zur Verfügung gestellt. Der Transport der Triosephosphate und der Import des Phosphats sind energieabhängig und verlaufen unter Mitwirkung eines Phosphat-Translokators.
