Botanik online 1996-2004. Die Seiten werden nicht mehr bearbeitet, sie bleiben als historisches Dokument der botanischen Wissenschaft online erhalten!
Wie alle molekularen Mechanismen ist Rekombination ein komplexer Vorgang, der in mehreren Teilabschnitte abläuft. Dazu gehören:
Da Spezifitäten im Spiele sind, ist zu fordern, daß Katalyatoren beteiligt sind, und das wiederum heißt, daß es rekombinationsdefekte Mutanten geben muß. Bei dem Bakterium Escherichia coli gibt es mindestens vier Genorte, deren Genprodukte an der Rekombination mitwirken: recA, recB, recC und lex. Das recA - Genprodukt ist unerläßlich. Mutationen an diesem Genort senken die Rekombinationshäufigkeit um fünf Größenordnungen herab und verursachen schwere physiologische Schäden. Die Empfindlichkeit gegenüber UV- und Röntgenstrahlung sowie alkylierenden Reagentien steigt an, die Induzierbarkeit von Prophagen und die UV-induzierte Mutationsrate sinken ab. Das recA -Genprodukt ist ein Protein mit dem Molekulargewicht 43.000 (McENTEE et al., 1976).
Es beeinflußt die Reparatur von DNS und greift in die DNS-Synthese regulierend ein. Die Genprodukte von recB, recC und lex sind Untereinheiten von Exo- und Endonukleasen, die auch an Reparaturmechanismen der DNS beteiligt sind. Diese Befunde weisen bereits darauf hin, daß Rekombination und Reparatur manches gemeinsam haben und in der Tat als zwei Seiten einer Medaille anzusehen sind.
Der Bakteriophage T4 besitzt ein "Gen 32-Protein", das von HUBERMAN et al. 1971 aus T4-infizierten Zellen isoliert wurde und dem die Eigenschaft eines Unwinding-Proteins (Entwindungsprotein; "Reißverschluß-Protein") zugeschrieben werden konnte. Nach Zugabe dieses Proteins zur DNS wird die Schmelztemperatur um etwa 20°C abgesenkt. Das Protein stabilisiert die Trennung von Doppelsträngen. Und das wiederum fördert nicht nur die Replikation, sondern auch die Rekombination.
Proteine mit gleicher oder sehr ähnlicher Wirkung sind inzwischen auch bei einer Reihe weiterer ein- und vielzelliger Organismen entdeckt worden. R. HOLLIDAY (National Institute for Medical Research, Mill Hill, London) stellte 1964 ein Rekombinationsmodell vor, das im wesentlichen auch heute noch gilt. Demnach wird die Rekombination durch einen Einzelstrangbruch eingeleitet. Das entstandene freie Ende löst sich ab und verdrängt den homologen Strang im benachbart liegenden Doppelstrang. Als essentielles Intermediärprodukt entsteht ein Heteroduplex (eine hybride DNS). Anschließend werden homologe Einzelstränge kreuzweise (reziprok) ausgetauscht.
Modelle lassen sich auf dem Papier zwar ganz schön zeichnen, sind allerdings nur dann sinnvoll, wenn sich die dargestellten Prozesse auch strukturell realisieren lassen. N. SIGAL und B. ALBERTS bauten 1972 maßstabsgetreue Kalottenmodelle und demonstrierten damit, daß von dieser Seite her keine Bedenken zu erwarten seien. Der Überkreuzungspunkt ist keine statische Struktur. Er kann durch Diffusion entlang des Moleküls, einem Reißverschlußmechanismus gleich, wandern.
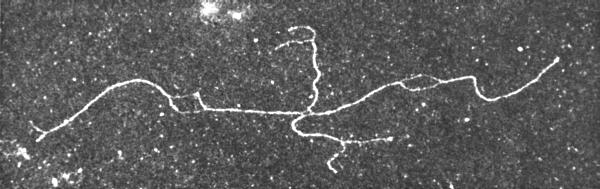
Eine elektronenmikroskopische Analyse brachte schließlich auch den optischen Beweis für die Existenz der Intermediärprodukte. H. POTTER und D. DRESSLER (Harvard University, 1976) untersuchten die Rekombinationseigenschaften des Plasmids ColE1 und bestätigten HOLLIDAYs Modell.

Elektronenmikroskopische Aufnahme eines rekombinationsintermediärprodukts: X - förmige Struktur zweier miteinander rekombinierender DNS-Moleküle, hergestellt in Anwesenheit hoher Formamidkonzentration. Unter diesen Bedingungen bleiben AT-reiche Abschnitte einsträngig. Diese sequenzspezifische Denaturierung erlaubt es, im Molekül homologe Arme zu identifizieren, darüberhinaus können die kovalenten Strangverknüpfungen im Bereich der Synapse verfolgt werden. Bei diesem wie auch bei 80 anderen Beispielen liegen die homologen Arme in trans-Konformation. (Aufn.: H. POTTER und D. DRESSLER, 1976)
Da inzwischen alle notwendigen molekularen Komponenten bekannt sind, konnte R. BERGMANN (Universität Hamburg, 2002) ein Chime Tutorial entwickeln, das die einzelnen Schritte beschreibt und den Vorgang der Rekombination auf molekularer Ebene veranschaulicht.
Bruch-Fusions-Brücken; Kontrollelemente; instabile (variable)
Gene
Seit Beginn des Jahrhunderts ist bekannt, daß es bei Pflanzen instabile oder variable Genloci gibt; man konnte sich jedoch zunächst nicht die dort drastisch gesteigerte Mutabilität und die erhöhte Rückmutationsrate erklären. Den entscheidenden Durchbruch erzielte B. McCLINTOCK durch ihre 1947 und 1951 veröffentlichten Untersuchungen an Maischromosomen. Die Grundlagen hierzu lieferten ihre früheren Beobachtungen und Analysen (1938) von Bruch-Fusions-Brücken, deren Auftreten mit Umstrukturierungen in Chromosomen korreliert werden konnte.
Brücken treten in der Anaphase immer dann in Erscheinung, wenn zwei Chromosomen an ihren Enden miteinander fusionieren und dadurch ein Fusionsprodukt mit zwei Centromeren entsteht. Werden die beiden nunmehr in entgegengesetzte Richtungen gezogen, kommt es zwangsläufig zu einem Chromosomenbruch. In der folgenden S-Phase des Interphasekerns wird ein Chromatid mit dem Bruch am Ende ebenso wie die übrigen repliziert, wodurch es zu einer erneuten Fusion der homologen Chromatiden kommt. In der anschließenden Mitose erscheint, statt eines Chromosoms aus zwei Chromatiden und einem Centromer, eines mit nur einem Chromatid, aber zwei Centromeren. Die Folge davon ist ein erneuter Bruch in der Anaphase, und von da ab geht der Zyklus in eine neue Runde.
 B.
McCLINTOCK erkannte, daß die Bruchstelle nicht an beliebiger Stelle
entlang der Chromosomenachse lag, sondern auf bestimmte Bereiche beschränkt
war, die sie Ds (dissociation) nannte. Offensichtlich handelt es sich dabei
um Abschnitte (wie später gezeigt wurde, DNS-Stücke), die zur
Entstehung von Translokationen, Deletionen, Inversionen, zur Bildung von
Ringchromosomen u.a. beitragen.
B.
McCLINTOCK erkannte, daß die Bruchstelle nicht an beliebiger Stelle
entlang der Chromosomenachse lag, sondern auf bestimmte Bereiche beschränkt
war, die sie Ds (dissociation) nannte. Offensichtlich handelt es sich dabei
um Abschnitte (wie später gezeigt wurde, DNS-Stücke), die zur
Entstehung von Translokationen, Deletionen, Inversionen, zur Bildung von
Ringchromosomen u.a. beitragen.
Der primäre Bruch verursacht in den Mitosezyklen der folgenden Generationen, während der Entwicklung zu verschiedenen Zeiten und an verschiedenen Stellen, gleichartige Brüche.
Der Abschnitt Ds, ein Mutatorgen, verhält sich so wie ein multiples Allel (besser Pseudoallel), das an verschiedenen Genorten lokalisiert sein kann und keineswegs immer die gleiche Struktur hat. Bemerkenswerterweise kann dieser Mutator sich inmitten anderer Gene ansiedeln und sie so außer Funktion setzen (inaktivieren). Demnach ist er ein Kontrollelement, das seinen Ort im Chromosom wechselt, das springt oder wandert und überall dort, wo es hinkommt, Mutationen hervorruft (synonyme Bezeichnungen: springende Gene, jumping genes).
Bald zeigte sich, daß es noch einen Satz weiterer Elemente geben mußte: Ac (activation) und daß ein Chromosomenbruch oder eine Translokation von Ds der Mithilfe von Ac bedarf. Ac kann ebenfalls als ein multiples Allel angesehen werden; es kann an den verschiedensten Stellen in allen Chromosomen auftreten. Um seine Wirkung genauer zu analysieren, konzentrierte sich B. McCLINTOCK auf das Studium von Genen, die das Farbmuster der Maiskörner determinieren.
Eines der wichtigsten ist der C-Locus, der in dominantem Zustand eine dunkelrote Färbung von Aleuronschicht und Perikarp (Samenschale) des Maiskorns hervorruft. Springt ein Ds-Element in das Gen hinein, unterbleibt die Farbstoffsynthese und es entstehen farblose (gelbliche) Körner. Eine Ac-Aktivität in ihnen ruft ein Muster dunkelroter Bereiche auf hellem Grund hervor. Die Erklärung hierfür liegt in einer Wiederherstellung des alten Zustands, weil durch das Ac-Element das Ds aus dem C-Locus wieder entfernt wird. Dieser Vorgang spielt sich während der Maiskornentwicklung in einigen der Zellen ab, aus denen sich die Aleuronschicht und das Perikarp entwickeln, und nur in den Klonen, die aus den veränderten Zellen durch Teilung entstehen, ist die Rückmutation sichtbar
 Heute
sind eine Reihe von Genloci bekannt, die durch das Ds-Ac-System oder andere
Kontrollelemente beeinflußt werden. Die Entdeckung des Spm-Systems
(supressor-mutator) und die Aufklärung seiner Funktion ergab, daß
die Kontrollelemente nicht allein ja/nein-Entscheidungen treffen, sie also
nicht nur Schalterfunktionen ausüben, sondern daß sie auch den
Grad der Genexpression modulieren.
Heute
sind eine Reihe von Genloci bekannt, die durch das Ds-Ac-System oder andere
Kontrollelemente beeinflußt werden. Die Entdeckung des Spm-Systems
(supressor-mutator) und die Aufklärung seiner Funktion ergab, daß
die Kontrollelemente nicht allein ja/nein-Entscheidungen treffen, sie also
nicht nur Schalterfunktionen ausüben, sondern daß sie auch den
Grad der Genexpression modulieren.
B. McCLINTOCKs genetische Analysen blieben jahrelang unverstanden. Erst als Ende der sechziger Jahre in bakterieller DNS Insertionselemente und Transposons gefunden und charakterisiert worden sind, zeichnete sich eine Analogie zwischen ihnen und den Kontrollelementen ab. Die vorliegenden genetischen Daten paßten widerspruchslos zu molekularbiologischen Modellvorstellungen (P. NEVERS und H. SAEDLER, 1977, H.-P. DÖRING und P. STARLINGER, 1984).
In Anerkennung ihrer Pionierleistung wurde 1983 der Nobelpreis für Medizin und Physiologie an Frau Barbara McCLINTOCK verliehen.
Anfang 1986 stellten P. NEVERS, N. S. SHEPHERD und H. SAEDLER eine Liste der in der Literatur beschriebenen "labilen Pflanzengene" auf. Aus ihr geht hervor, daß solche Gene bei über 30 Arten analysiert worden sind. Viele dieser Mutanten, mit Namen wie variegata, mutabilis, marmorata, maculata oder variabilis, sind wegen der unregelmäßigen Scheckungen ihrer Blüten oder Blätter als Sorten (Zierpflanzen) im Handel.
Ein Transposon ist ein Stück DNS, das von zwei gegenläufig
orientierten Insertionselementen (IS-Elementen) flankiert wird (ein Palindrom).

Aufbau eines Transposons (Schema): oben: im DNS-Doppelstrang, unten: durch Denaturierung des Doppelstrangs (man beachte: nur einer der Stränge ist hier gezeichnet). Die invers orientierten IS-Elemente (Insertionselemente) an den Transposonenden bilden einen doppelsträngigen Abschnitt.
Die IS-Elemente beeinflussen die Expression benachbarter Gene, können Deletionen induzieren und haben darüber hinaus die Eigenart, sich in das Bakteriengenom ein- und wieder auszubauen. Durch diesen Ein- und Ausbau können die auf einem Transposon liegenden, von IS-Elementen flankierten Gene, mit übertragen (transponiert) werden. Ein Ein- und Ausbau erfolgt aber nur dann, wenn die Zelle die dafür benötigten Enzyme bereitstellt. Dies wiederum heißt, daß für sämtliche Strukturveränderungen der DNS und der Chromosomen (Deletionen, Duplikationen. Inversionen, Insertionen usw.) zwei Komponenten erforderlich sind, nämlich einmal die Enzyme, zum anderen bestimmte Erkennungssequenzen in der DNS: die Transposons. Das heißt aber auch, daß diese Prozesse zwar an vielen Orten entlang des DNS-Fadens auftreten können, aber nicht an jeder beliebigen Stelle (site-specific recombination). Am Erkennungsprozeß sind komplementäre Nukleotidsequenzen beteiligt, die an verschiedenen Stellen im Genom verstreut sind; der Austausch der genetischen Information erfolgt über einen Rekombinationsprozeß. Doch im Gegensatz zu den Vorgängen während der Meiose, bei der sich die homologen Chromosomen entlang ihrer ganzen Länge paaren, kommt es hier nur zu einer Paarung relativ kurzer Abschnitte. Man spricht daher (vielleicht nicht ganz korrekt) von illegitimem Crossing-over.

Das Ausschneiden von Abschnitten aus einem RNS-Molekül wird Gene-splicing genannt. Zum Nachweis werden die RNS-Stränge mit den entsprechenden Genabschnitten der DNS (nachdem diese geschmolzen, und damit in Einzelstränge zerlegt wurde) zusammengegeben. Es bildet sich ein mRNS-DNS-Hybrid, bei dem eine DNS-Schlaufe ausgespart bleibt (H. WESTPHAL und S. P. LAI, Bethesda, 1977)
Der Analogiebeweis zwischen den McCLINTOCKschen Kontrollelementen und den bakteriellen Transposons wurde durch deren molekularbiologische Analyse (Isolierung, Klonierung der DNS-Abschnitte in Bakterien und anschließende Sequenzierung) erbracht.
Die Mais-Transposons treten oft in Paaren auf, wobei eines der Elemente autonom ist, d.h., alle ihm eigenen Funktionen selbst ausüben kann, während das andere passiv ist und nur bei Anwesenheit des aktiven Elements aktiviert oder mobilisiert werden kann (wie für das Ac-Ds-Paar bereits besprochen).
Ähnliche Verhältnisse findet man bei Drosophila. Dort zeigte sich, daß sich das aktive Element vom passiven lediglich durch eine interne Deletion unterschied. Es lag daher nahe, auch die Ds- und Ac-Nukleotidsequenzen auf einen derartigen Unterschied hin zu untersuchen. N. FEDEROFF (Carnegie Institution of Washington) zeigte durch Hybridisierungsversuche mit isolierter Ds- und Ac-DNS, daß sich erstere tatsächlich durch das Fehlen eines internen, ca. 200 Basenpaare langen Abschnitts von der zweiten unterscheidet. Das analysierte Ac-Element bestand aus 4300 Basenpaaren, das entsprechende Ds-Element aus 4100. Ein anderes der untersuchten Ds-Elemente (Allele) enthielt nur 1700 Basenpaare.
Es sind mittlerweile eine ganze Reihe unterschiedlich langer Ds-Elemente isoliert und zum Teil auch sequenziert worden. Das kürzeste (Ds 1) ist nur 405 Basenpaare lang (von J. C. OSTERMAN und D. SCHWARTZ isoliert, 1981). Aus Mutanten mit defektem Sucrosesynthetasegen (Saccharosesynthetasegen) [dem Sh-(shrunken-) Locus] isolierten P. STARLINGER und Mitarbeiter (Genetisches Institut der Universität zu Köln) eine über 30 000 Basenpaare lange Insertion, die an beiden Enden von Ds-Elementen flankiert ist

Insertion im Shrunken-Locus des Mais. Das Gen codiert die Sucrosesynthetase (eines der wichtigsten Proteine im Endosperm). In einer Mutante ist es durch Insertion eines ca. 30 000 Basenpaare langen Abschnitts inaktiviert. Dieser Abschnitt ist von Ds-Elementen in unterschiedlicher Orientierung flankiert. Da die Enden des gesamten Abschnitts einander komplementäre Sequenzen tragen (< >), können sich diese unter Mitwirkung eines Enzyms (Transponase) untereinander paaren, eine Transposonstruktur ausbilden, und aus dem Sucrosesynthetasegen wieder ausgeschnitten werden. Das Ausschneiden kann aber auch an anderer Stelle erfolgen, sofern einander komplementäre Sequenzen vorhanden sind (< > oder > <). Das Modell veranschaulicht, daß der Abschnitt zwischen den Ds-Elementen für das Ausschneiden (und das spätere Einsetzen an anderer Stelle) belanglos ist. Ein vollständiges Gen beispielsweise kann übertragen werden, wenn es an beiden Enden Ds-Elemente trägt. (Nach P. STARLINGER, 1985)
Bemerkenswert ist dabei der Befund, daß jedes der Enden zwei vollständige, respektive ein vollständiges und ein unvollständiges Ds-Element in entgegengesetzter Orientierung tragen. Die Nukleotidsequenzen an den Enden von Ds-Elementen sind stets
T-A-G-G-A-T-G-A-A.
In Ac-Elementen kann das terminale T durch ein C ersetzt sein. Zwei der in Köln sequenzierten Ac-Elemente sind 4563 Basenpaare lang.
Wie bereits dargelegt, sind Kontrollelemente keineswegs auf Mais beschränkt. H. SAEDLER und Mitarbeiter (Max-Planck-Institut für Züchtungsforschung, Köln) analysieren deren molekulare Struktur bei Antirrhinum majus, andere untersuchen die der Petunien. Die Erkenntnis, daß es im Pflanzenreich Kontrollelemente gibt, die sich wie Transposons verhalten, bietet eine glaubwürdige (und eines Tages auch von Fall zu Fall überprüfbare) Erklärung für eine Fülle von bislang wenig beachteten oder unerklärten Phänomenen. Eine Musterbildung, z.B. eine unregelmäßige Farbverteilung in Blüten, oder eine ungleiche Verteilung von chlorophyllhaltigen und chlorophyllfreien Bereichen in Blättern, sind der sichtbare Ausdruck sprunghafter Veränderungen von Genaktivitäten im Verlaüf der Ontogenese. Diese Veränderungen folgen aber nicht einem starren, reproduzierbaren Entwicklungsprogramm, wie z.B. bei den Sektorial- oder Periklinalchimären, sondern treten zufallsgemäß (statistisch) auf. Mit der Annahme, daß hier Kontrollelemente oder Kontrollelementsysteme (einschließlich der für die Übertragung notwendigen Enzyme) am Werk sind, können wir die Erscheinungen widerspruchslos deuten. Neben ihrer Bedeutung für die Ausprägung von Merkmalen im Verlauf der Ontogenese spielen sie vermutlich auch eine entscheidende Rolle bei der Beschleunigung von Evolutionsprozessen. Wie wir später noch sehen werden, unterscheiden sich die Genome verwandter Arten oft durch den Ploidiegrad, durch Chromosomenumstrukturierungen, Reduktion oder Vervielfachung von Heterochromatin, oder durch Aktivitätsverlust einzelner Gene. Daraus ist zu schließen, daß die Evolution vielzelliger, eukaryotischer Organismen weniger auf der Akkumulation von Punktmutanten als vielmehr auf Umstrukturierung vorhandener genetischer Information beruht. Das Wissen über die Kontrollelemente / Transposons lehrt uns, wie diese Veränderungen auf molekularer Ebene vor sich gehen (können).