Botanik online 1996-2004. Die Seiten werden nicht mehr bearbeitet, sie bleiben als historisches Dokument der botanischen Wissenschaft online erhalten!
Unter Transkription versteht man jenen Vorgang, bei dem einer der beiden DNS-Stränge als Matrize zur Bildung von RNS verwendet wird. Die Information wird demnach von DNS auf RNS überschrieben, wobei jedoch zu vermerken ist, daß die in der RNS und die im Matrizenstrang der DNS enthaltenen Basensequenzen einander komplementär sind.
Bei der Transkription werden immer nur Teilinformationen abgerufen. Lediglich bei einigen kleinen Viren, deren Genom größenordnungsmäßig drei Gene trägt, kann die Transkription en bloc erfolgen.
Katalysiert wird die Transkription durch DNS-abhängige RNS-Polymerasen. Die Enzyme aus Prokaryoten unterscheiden sich in entscheidenden Merkmalen von denen aus Eukaryoten. Aufgrund unterschiedlicher Funktion kann man die Transkriptionsprodukte (Transkripte) drei Klassen zuordnen:
![]() mRNS (messenger RNS)
mRNS (messenger RNS)
![]() tRNS (transfer RNS)
tRNS (transfer RNS)
![]() rRNS (ribosomale RNS)
rRNS (ribosomale RNS)
Bei Eukaryoten entsteht - anstelle der mRNS zunächst im Zellkern eine weniger klar definierte Klasse, die hnRNS (heterogene RNS), deren überwiegender Teil unmittelbar nach der Synthese wieder abgebaut wird. Ein nur geringer, durch Teilab- und -umbau verbleibender Anteil wird über einen komplexen, aber hochspezifischen Prozeß (Processing) zu mRNS verarbeitet. RNS nimmt in der Zelle viele Funktionen wahr. Seit einigen Jahren sind Verfahren zur Bestimmung von Nukleotidsequenzen in Anwendung, und viele RNS-Sorten wurden analysiert. Aus bekannten Sequenzen ist ablesbar
| wie ein RNS-Molekül gefaltet ist (d.h. welche Sekundärstruktur und Tertiärstruktur es einnimmt), | |
| welche Bereiche mit Proteinen oder anderen Nukleinsäuren in Wechselwirkung stehen, | |
| welche Beziehung zwischen einem Gen (einem Abschnitt in der DNS) und einem Transkriptionsprodukt besteht. Aus dem Vergleich folgt, welche Veränderungen die RNS durchgemacht hat, damit aus einer Vorstufe ein fertiges (gereiftes und funktionsfähiges) Produkt wird, | |
| wie hoch der Homologiegrad der RNS aus verschiedenen Organismen ist. Solche Angaben können zur Klärung von Verwandtschaftsbeziehungen der betreffenden Organismen herangezogen werden. |
mRNS trägt genetische Information, d.h., die Instruktion zur Synthese einer (bei Prokaryoten oft auch mehrerer) Polypeptidketten. Fertige mRNS der Eukaryoten enthält zwischen 400 und 4000 Nukleotidbasen. Anfang und Ende tragen zusätzliche, nicht durch den Transkriptionsprozeß erworbene Sequenzen. Am 5'-Ende findet man bei vielen, doch lange nicht bei allen im Cytosol vorliegenden mRNS-Molekülen eine "Kappe" (capping), ein spezifisches Oligonukleotid, in dem die Basen in einer in Nukleinsäuren sonst nicht üblichen Verknüpfung untereinander verbunden sind; am 3'-Ende kommt bei 30-40 Prozent aller aus dem Cytosol isolierten mRNS-Moleküle eine Poly-A-Sequenz vor (Länge bis zu 200 Nukleotiden). Nur ein Teil der fertigen (reifen) mRNS wird translatiert. Am Anfang, der Kappe folgend, steht eine nicht codierende Sequenz (Leadersequenz; Länge: größenordnungsmäßig 10-200 Nukleotide). Die codierende Sequenz beginnt mit dem Initiatorcodon AUG und endet mit einem der drei Terminatorcodons (UAG, UAA oder UGA).
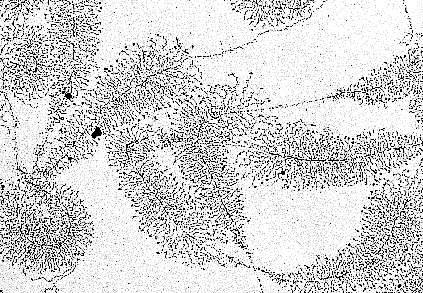
In den Nukleoli der Oozytenkerne von Triturus virescens, einer amerikanischen Molchart (sowie bei anderen Amphibien) liegt DNS frei vor. In der Abbildung ist die Transkription der Gene sichtbar, die die Information zur Bildung ribosomaler RNS tragen ("MILLER-Bäumchen"). Mehr zur Deutung des elektronenmikroskopischen Bildes (O. L. MILLER, B. R. BEATTY, Biology Division, Oak Ridge National Laboratory, 1969)
Ihm folgt eine weitere, nicht codierende Sequenz, deren Länge bis zu 600 Nukleotide betragen kann. Es sind bisher erst relativ wenige pflanzliche Nukleinsäüreabschnitte (Gene, nicht codierende Bereiche) sequenziert worden. Bekannt ist u.a. die Nukleotidabfolge im Gen für die kleine Untereinheit der Ribulose-1,5-Bisphosphatcarboxylase. Die Länge beträgt 668 Nukleotide, davon machen 440 die codierende Sequenz aus.
tRNS stellt eine Klasse relativ kleiner Moleküle dar. Sie enthalten 70-90 Nukleotidbasen. Viele von ihnen sind modifiziert und gehören damit in die Kategorie der seltenen Basen. Die Modifikation erfolgt im Anschluß an die Transkription unter Mitwirkung spezifischer Enzyme. tRNS-Moleküle falten sich in sich selbst auf und bilden damit eine charakteristisch aussehende Sekundärstruktur (Kleeblattstruktur) aus. Eine weitere Faltung führt zu einer definierten Tertiärstruktur. An ihrer Stabilisierung sind die seltenen Basen beteiligt. Zwei Funktionen sind der tRNS zuzuschreiben:
| erstens die Erkennung eines Codons in der mRNS durch Basenpaarung (Codon-Anticodon), | |
| zweitens die Erkennung der "dazugehörigen" Aminosäure. Das geschieht unter Mitwirkung einer Gruppe von Enzymen, den Aminoacyl-tRNS-Synthetasen (auch Aminosäure-aktivierende Enzyme genannt). |

Nebenstehendes Bild: Seryl-tRNS-Synthetase, komplexiert mit tRNS. (vom tRNS-Molekül sind - durch violette Kugeln symbolisiert - nur die Phosphatreste abgebildet).
© J. Sühnel, Image Library, Jena
Wie viele verschiedene tRNS-Moleküle gibt es in einer Zelle? Die theoretische Mindestzahl wäre 20, das würde der Anzahl verschiedener, in Proteine eingebauter Aminosäuren entsprechen. Der Maximalwert wäre 61 (= 64 - 3), denn für die drei Stoppcodons gibt es keine tRNS. Die tatsächliche Zahl liegt etwa in der Mitte. Für eine Anzahl von Aminosäuren sind mehrere verschiedene tRNS-Arten nachgewiesen wobden, doch kann eine tRNS oftmals mehrere Codons erkennen, wenn sie sich lediglich in der dritten Position voneinander unterscheiden (wobble).
Es kommt dann zu inkorrekten, thermodynamisch dennoch ausreichend stabilen Paarungen. Es genügt also, wenn an der dritten Position zwischen einem Purin und einem Pyrimidin unterschieden wird (siehe hierzu auch die Tabelle der Codeworte).
In Zellen grüner Pflanzen kommt eine weitere Komplikation hinzu: Sowohl Kern als auch Chloroplasten und Mitochondrien enthalten genetische Information, und in jedem der Kompartimente findet eine von den anderen weitgehend unabhängige Genexpression (Transkription, Translation) und Replikation statt. Lediglich über Kontrollmechanismen werden die Prozesse in den einzelnen Kompartimenten aufeinander abgestimmt. Sowohl die Mitochondrien als auch die Chloroplasten verfügen über einen eigenständigen Satz an tRNS, und die dafür erforderliche genetische Information liegt in der DNS der Mitochondrien bzw. Chloroplasten.
Die ribosomale RNS ist eine Strukturkomponente der Ribosomen. Sowohl die kleine als auch die große Untereinheit enthalten je ein, respektive zwei (drei) unterschiedlich große rRNS-Moleküle. Die Größe wird meist durch den S-Wert (die Sedimentationskonstante) angegeben. Wiederum findet man auch bei dieser RNS-Klasse unterschiedliche Sätze im Cytosol, den Chloroplasten und den Mitochondrien.
rRNS-Moleküle in Ribosomen des Cytosols sind beträchtlich größer als die entsprechenden in den Chloroplasten-Ribosomen. Letztere sind in der Größe mit rRNS aus Prokaryoten vergleichbar. Die Nukleotidsequenzen einer Anzahl von rRNS-Typen unterschiedlicher Herkunft sind bekannt, und die Ergebnisse lassen sich zu Verwandtschaftsstudien heranziehen. So bilden gerade diese Daten eine der sichersten Stützen der Endosymbiontenhypothese. Hilfreich ist die Tatsache, daß rRNS, wie die tRNS, Sekundärstrukturen ausbildet, die jedoch wesentlich komplizierter aussehen. Es besteht eine weitgehende Übereinstimmung solcher Strukturen der rRNS aus Chloroplasten und aus Blaualgen.
Die genetische Information zur Instruktion der rRNS (in Chloroplasten) wird in einem Stück transkribiert. Durch nachfolgendes Processing wird das Transkript in 23 S, 5 S, 4,5 S und 16 S rRNS zerlegt.
Vergleichbare Mechanismen liegen auch der Synthese anderer RNS-Arten (auch in anderen Kompartimenten) zugrunde. Weitere Daten über rRNS siehe rRNA-www-Server
Weit mehr als bei Prokaryoten-RNS, unterliegt die der Eukaryoten im Anschluß an die Transkription einem umfangreichen Processing. Zwei der Veränderungen wurden bereits besprochen:
|
| Capping und Polyadenylierung von mRNS |
| Bildung seltener Basen in tRNS. |
Hinzu kommt, daß die primären Transkripte durchweg länger als die fertigen Produkte sind. Auch hierfür wurde schon ein Beispiel angeführt. Eine Anzahl von Genen besteht aus mehreren Segmenten. Die translatierbaren, codierenden Abschnitte bezeichnet man als Exons (Abschnitte, die exprimiert werden), die dazwischenliegenden als Introns. Das primäre Transkriptionsprodukt enthält beides: Exons, die durch Introns voneinander getrennt sind.
Vor einer Translation müssen letztere herausgeschnitten werden. Es gibt eine Anzahl spezifischer Ribonukleasen, und zumindest einem Teil von ihnen fällt die Aufgabe zu, primäre Transkripte (hnRNS) in die funktionelle Form (z.B. mRNS, aber auch tRNS, rRNS) zu überführen. Der überwiegende Teil der im Kern gebildeten hnRNS erreicht nie das Cytosol, sondern wird unmittelbar im Anschluß an die Synthese wieder degradiert. Es gibt zwar noch keine schlüssige Erklärung für diese hohe Umsatzrate, doch wird als eine mögliche Erklärung hierfür die Annahme genannt, daß Ribonukleosidtriphosphate in großer Menge auf Abruf bereitgehalten werden müssen. In hoher Konzentration würden sie jedoch einen beträchtlichen osmotischen Druck hervorrufen. Durch Polymerisation könnte dieser auf physiologische Werte gesenkt werden.
Im Cytoplasma liegt mRNS nie in freier Form vor, sondern ist stets an spezifische Proteine gebunden und formt so einen Ribonukleoproteinkomplex (RNP).
Die DNS im Zellkern ist in der Regel hochgradig kondensiert. Daher ist es nicht ohne weiteres möglich, diese Transkriptionseinheiten im Elektronenmikroskop abzubilden. Es gibt jedoch eine Gruppe von Zellen mit Lampenbürstenchromosomen, bei denen die DNS im Kern aufgelockert ist. Von einer chromosomalen Zentralachse ausgehend, sind eine Vielzahl von Schlaufen "ausgefahren". Lampenbürstenchromosomen findet man z.B. in den Oozyten von Amphibien, doch auch in Zellen der Grünalge Acetabularia mediterranea. An der freiliegenden DNS können Transkriptionseinheiten nachgewiesen werden. Durch Auswertung elektronenmikroskopischer Bilder sind folgende Schlüsse zu ziehen:
Transkriptionseinheiten können unterschiedlich lang sein; es gibt aber auch gleichartige, die tandemartig hintereinandergeschaltet sind.
Es kommen Transkriptionseinheiten mit entgegengesetzten Polaritäten vor. Das beruht darauf, daß einmal der eine, im anderen Fall der andere DNS-Strang genutzt wird. Entscheidend ist allein die Lage des Promoters, also jener Nukleotidabfolge, die für die Bindung der DNS-abhängigen RNS-Polymerase benötigt wird und die den Startpunkt der Transkription bildet.
Zwischen den Transkriptionseinheiten liegen unterschiedlich lange, nicht transkribierte Spacer (Lücken, Abstandshalter).
Die Länge der Transkripte erscheint wesentlich kürzer als die der Matrize. Die Ursache hierfür ist darin zu suchen, daß die sich bildende RNS unmittelbar nach der Synthese Sekundärstrukturen (Palindrome = Haarnadelstrukturen) ausbildet und/oder mit Proteinen Komplexe eingeht, so daß sich die scheinbare Moleküllänge (Konturlänge) drastisch verkürzt.
Es gibt Gene mit hoher, und solche mit geringer Transkriptionsrate.
Polymerasen sind zunächst einmal, allgemein gesagt, Enzyme, die für die Bildung von Polynukleotiden benötigt werden. Die meisten von ihnen sind DNS-abhängig, d.h. sie benötigen eine Matrize. DNS-abhängige RNS-Polymerasen erkennen auf der DNS Abschnitte (Promotoren), werden dort gebunden und leiten an den Stellen einen Transkriptionsvorgang ein. Er endet, sobald die Polymerase eine Terminationssequenz erreicht (und dort von der DNS abfällt). Repressoren sind Proteine, die (in aktivem Zustand) fest an DNS binden, und damit die Transkription in dem Bereich unterdrücken. Die o.g. Polymerasen der Pro- und Eukaryoten unterscheiden sich grundsätzlich voneinander.
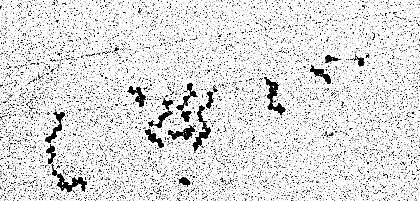
Transkription und Translation an einem DNS-Abschnitt von Escherichia coli. Die DNS ist als Faden erkennbar. Er wird durch mehrere DNS-abhängige RNS-Polymerase-Moleküle gleichzeitig transkribiert. Im Bild ist die Transkriptionsrichtung von rechts nach links an zunehmender Länge der sich bildenden mRNS zu erkennen, die in Form seitlicher Abzweigungen erscheint. Sobald Teile von ihr gebildet worden sind, heften sich Ribosomen an sie. Mit wachsender Länge steigt die Anzahl der gebundenen Ribosomen, an denen (ebenfalls wie am Fließband) Protein gebildet wird (D. L. MILLER, Charlottesville, 1970)
Kern-DNS wird durch drei verschiedene Klassen von RNS-Polymerasen transkribiert. Alle bestehen aus mehreren Untereinheiten (Polypeptidketten). In vielen Fällen sind zumindest die Molekulargewichte bekannt. Allein diese Werte weisen auf eine beträchtliche Variabilität hin. Sie machen deutlich, daß sich diese Proteine im Verlauf der Evolution nicht unwesentlich verändert haben. Von Klasse zu Klasse (Polymerase I, II, III) sind kaum Gemeinsamkeiten auszumachen.
Unabhängig von diesen drei Enzymen kommt in Chloroplasten ein weiterer Typ vor. Dieses Enzym hat gewisse verwandtschaftliche Beziehungen zum entsprechenden Prokaryotenenzym. Es sei aber erwähnt, daß es durch ein im Kern lokalisiertes Gen codiert wird.
In Analogie hierzu darf man wohl annehmen, daß es auch ein entsprechendes Enzym in den Mitochondrien gibt. Pflanzenzellen wurden daraufhin zwar noch nicht untersucht, doch wurde in Mitochondrien der Hefe und der tierischer Zellen ein solches Enzym nachgewiesen.
Untersuchungen an Escherichia coli haben ergeben, daß eine Transkriptionseinheit (ein Operon) außer der Matrize für die Transkripte eine Anzahl von Signalen enthält. Es gibt auf der DNS Start- und Stoppsignale, die den Anfang und das Ende einer Transkriptionseinheit (eines Operons) markieren. Es gibt darüber hinaus Bindungsstellen für Proteine, die die Transkription bestimmter Abschnitte fördern, hemmen, oder sogar blockieren.
Die Signalwirkung beruht in der Regel auf einer Wechselwirkung zwischen dem Protein (je nach Funktion: Aktivator oder Repressor) und einer definierten Nukleotidsequenz. Manche der Proteine können alternativ in einem aktiven oder inaktiven Zustand vorliegen.
Viele Promoterregionen eukaryotischer Gene enhalten etwa 25 - 32 Basenpaare vor dem eigentlichen Initiationspunkt der Transkription eine extrem konservative, an AT-Paaren reiche Sequenz, die als TATA-Box bekannt ist und die als Bindungsstelle für einen Transkriptionsfaktor (TATA-Box-Bindeprotein) dient. Eine spezifische Bindung dieses Proteins sichert eine korrekte Positionierung der RNS-Polymerase II, die für proteincodierende Gene benötigt wird, auf der DNS
Der Übergang kann durch Metaboliten oder extern zugeführte Nährstoffe (z.B. bestimmte Zucker) hervorgerufen werden. Vergleichbare Regelmechanismen werden auch für Eukaryoten postuliert, doch konnte bislang noch kein Mechanismus im Detail ausgearbeitet werden, wie wir ihn für einige bakterielle Operons kennen. Einige weitere Einzelheiten werden wir an anderen Stellen, so bei der Besprechung der Differenzierung, der Photomorphogenese und der Hormonwirkungen kennenlernen.
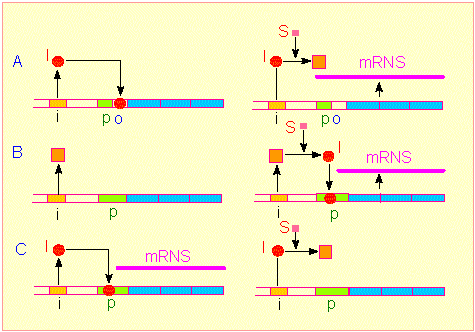
Drei unterschiedliche Kontrollmechanismen der Genregulation. Man unterscheidet zwischen Regulatorgenen (ocker) und Strukturgenen (hellblaz). Einer Gruppe von Strukturgenen (einem Operon) ist ein Promotor (grün: Startstelle der DNS-abhängigen RNS-Polymerase) und ein Operator (o: Bindungsstelle eines Regulatormoleküls [Induktor oder Repressor]) vorgeschaltet. Oberes Bildpaar: Substratinduzierte Kontrolle der Transkription. Der vom Regulatorgen codierte Repressor bindet an o und verhindert damit die Transkription der Strukturgene. Er ist durch ein Substrat inaktivierbar, damit wird die Transkription (gebildete RNS: violette Linie) der Strukturgene freigegeben. Die durch die Strukturgene codierten Enzyme sind am Abbau des genannten Substrats beteiligt (JACOB-MONOD-Modell). Mittleres Bildpaar: Vom Regulatorgen wird ein inaktiver Induktor codiert. Die Transkriptionsrate der Strukturgene ist gering. Durch ein Substrat wird der Induktor aktiviert, er bindet an den Promotor und fördert damit die Aktivität der DNS-abhängigen RNS-Polymerase, die Transkriptionsrate steigt. Unteres Bildpaar: Vom Regulatorgen wird ein aktiver Induktor codiert, die Transkriptionsrate der Strukturgene ist hoch. Durch Bindung eines Substratmoleküls wird der Induktor inaktiviert.